如果你手上有一個神奇基因清單,你想問一下有沒有神奇Pathway在裡面,首先你必需知道有多少基因被註解在已知的生物途徑中,最簡單的方法是用 [DAVID]
DAVID 內建 ID 轉換的功能,支持大部份的 ID ,解決了大部份使用者最棘手的ID轉換問題,經轉換後就成為DAVID ID,可以作後續的分析了(有關如何轉換ID請參閱)。
你要做的只是把基因清單貼上:
##ReadMore##
然後按下[Functional Annotation Tool],DAVID會使用GO analysis 的方法,計算每個Pathway的p 值,並用p 值篩選輸出Pathway
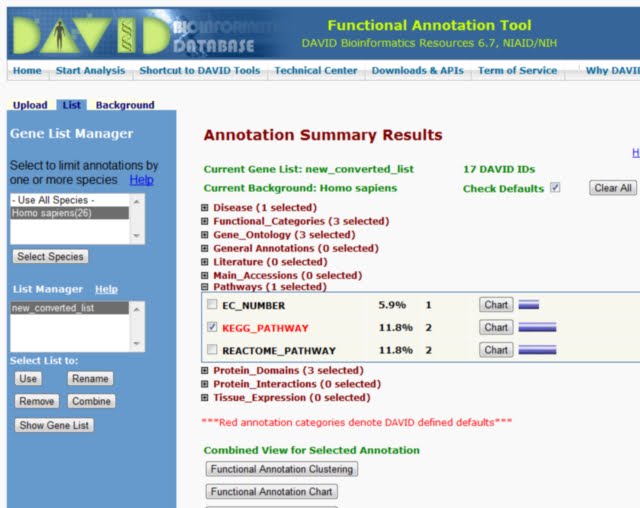
點選 [Pathway]展開 ,再按下[Chart]就會顯示結果,不過你可能會發現一個Pathway也沒有,因為如同<前文>所說的,這樣的p-value計算有些問題,所以我通常忽略這個篩選步驟。
方法昰按上面的[option],把 EASE門檻設為 1,就可忽略統計分析,而前面的選項是設定有多少基因在同一個pathway中出現,才顯示出來 再按[Return Using Options],符合條件的pathway就會顯示出來。
你會發現絕大部份時間,pathway中只有少部份的基因被標示出來,這便是我說pathway 不適合用GO analysis 的方法分析的原因,在pathawy中有太多是透過非表現量的方式調控,就算這個pathway真得出現在這實驗系統中,也未必能從p-值的估算辨識出來。所以通常只有被註解到的Pathway,都值得一看,反正也不會太多。
點選個別的pathway 就會出現在清單中的基因,在pathway 的位置了,很簡單吧!
DAVID 算是我最常用的註解工具,因為他可以線上的解決ID轉換的問題,而且速度也很快,唯一美中不足的是,他只能標示基因,不能把基因變化的趨勢(up/down)一起放在圖中,在Pathway中,有些基因是抑制,有些是促進,他們誰上誰下代表著截然不同的義意,如果我們可以所有基因表現變化標示在圖上,這樣可有助於我們思考整個pathway的運作。許多付費的軟體都可以作到這樣圖,免費的也有,例如Advance Pathway Painter or Cytoscape,如果你使用的KEGG資料庫。KEGG網頁上有內建這樣的功能,不過,這些軟體操作都有點複雜,改天單獨介紹。
DAVID 內建 ID 轉換的功能,支持大部份的 ID ,解決了大部份使用者最棘手的ID轉換問題,經轉換後就成為DAVID ID,可以作後續的分析了(有關如何轉換ID請參閱)。
你要做的只是把基因清單貼上:
##ReadMore##
然後按下[Functional Annotation Tool],DAVID會使用GO analysis 的方法,計算每個Pathway的p 值,並用p 值篩選輸出Pathway
點選 [Pathway]展開 ,再按下[Chart]就會顯示結果,不過你可能會發現一個Pathway也沒有,因為如同<前文>所說的,這樣的p-value計算有些問題,所以我通常忽略這個篩選步驟。
方法昰按上面的[option],把 EASE門檻設為 1,就可忽略統計分析,而前面的選項是設定有多少基因在同一個pathway中出現,才顯示出來 再按[Return Using Options],符合條件的pathway就會顯示出來。
你會發現絕大部份時間,pathway中只有少部份的基因被標示出來,這便是我說pathway 不適合用GO analysis 的方法分析的原因,在pathawy中有太多是透過非表現量的方式調控,就算這個pathway真得出現在這實驗系統中,也未必能從p-值的估算辨識出來。所以通常只有被註解到的Pathway,都值得一看,反正也不會太多。
點選個別的pathway 就會出現在清單中的基因,在pathway 的位置了,很簡單吧!
DAVID 算是我最常用的註解工具,因為他可以線上的解決ID轉換的問題,而且速度也很快,唯一美中不足的是,他只能標示基因,不能把基因變化的趨勢(up/down)一起放在圖中,在Pathway中,有些基因是抑制,有些是促進,他們誰上誰下代表著截然不同的義意,如果我們可以所有基因表現變化標示在圖上,這樣可有助於我們思考整個pathway的運作。許多付費的軟體都可以作到這樣圖,免費的也有,例如Advance Pathway Painter or Cytoscape,如果你使用的KEGG資料庫。KEGG網頁上有內建這樣的功能,不過,這些軟體操作都有點複雜,改天單獨介紹。





留言
張貼留言